install.packagesでのError
ちょっとハマったので備忘。
> install.packages("TTR") #(中略) clang -dynamiclib -Wl,-headerpad_max_install_names -undefined dynamic_lookup -single_module -multiply_defined suppress -L/usr/local/Cellar/r/3.2.1_1/R.framework/Resources/lib -L/usr/local/opt/gettext/lib -L/usr/local/opt/readline/lib -o xts.so add_class.o any.o attr.o binsearch.o coredata.o diff.o dimnames.o endpoints.o extract_col.o init.o isOrdered.o isXts.o leadingNA.o merge.o period.max.o period.min.o period.prod.o period.sum.o rbind.o rollfun.o runSum.o startofyear.o subset.o subset.old.o toperiod.o totalcols.o tryXts.o unique.time.o -L/usr/local/Cellar/gcc/5.2.0/lib -L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5/gcc/x86_64-apple-darwin14.3.0/5.1.0 -L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5 -lgfortran -lquadmath -lm -F/usr/local/Cellar/r/3.2.1_1/R.framework/.. -framework R -lintl -Wl,-framework -Wl,CoreFoundation ld: warning: directory not found for option '-L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5/gcc/x86_64-apple-darwin14.3.0/5.1.0' ld: warning: directory not found for option '-L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5' ld: library not found for -lgfortran clang: error: linker command failed with exit code 1 (use -v to see invocation) make: *** [xts.so] Error 1 ERROR: compilation failed for package ‘xts’ * removing ‘/usr/local/Cellar/r/3.2.1_1/R.framework/Versions/3.2/Resources/library/xts’ ERROR: dependency ‘xts’ is not available for package ‘TTR’ * removing ‘/usr/local/Cellar/r/3.2.1_1/R.framework/Versions/3.2/Resources/library/TTR’ ダウンロードされたパッケージは、以下にあります ‘/private/var/folders/b7/5pf80zmn03d5tjwx6zbd0jb400123gn/T/RtmxWfWtNZ/downloaded_packages’ 警告メッセージ: 1: install.packages("TTR") で: パッケージ ‘xts’ のインストールは、ゼロでない終了値をもちました 2: install.packages("TTR") で: パッケージ ‘TTR’ のインストールは、ゼロでない終了値をもちました
ということで、「libgfortranがないっす」って言われてるので探してみる。
$ mdfind -name libgfortran.a /usr/local/Cellar/gcc/5.2.0/lib/gcc/5/libgfortran.a /usr/local/Cellar/gcc/5.2.0/lib/gcc/5/i386/libgfortran.a
ありますねー、そしてこのディレクトリも-Lで既に入ってんじゃん、、って思ったら、-L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5が悪さしてそう・・・
なんだよなんだよ余計なもんつけやがって、、、で、今度はRのoption設定してるファイル探す。
#Rのディレクトリ漁ってたら怪しいファイル発見。 $ vi /usr/local/Cellar/r/3.2.1_1/R.framework/Versions/3.2/Resources/etc/Makeconf 1 # etc/Makeconf. Generated from Makeconf.in by configure.$ 2 #$ 3 # ${R_HOME}/etc/Makeconf$ 4 #$ 5 # R was configured using the following call$ 6 # (not including env. vars and site configuration)$ 7 # configure '--prefix=/usr/local/Cellar/r/3.2.1_1' '--with-libintl-prefix=/usr/local/opt/gettext' #(中略) 44 FFLAGS = -g -O2 $(LTO)$ 45 FLIBS = -L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5/gcc/x86_64-apple-darwin14.3.0/5.1.0 -L/usr/local/C 46 FCPICFLAGS = -fno-common$
ここにいたああああ!ということで、早速書き換える。
44 FFLAGS = -g -O2 $(LTO)$ 45 #FLIBS = -L/usr/local/Cellar/gcc/5.1.0/lib/gcc/5/gcc/x86_64-apple-darwin14.3.0/5.1.0 -L/usr/local/ 46 FLIBS = -L/usr/local/Cellar/gcc/5.2.0/lib/gcc/5/ -lgfortran -lquadmath -lm$
このあとインストールしたら無事できました。めでたしめでたし。
前処理
ということで、ようやく週末でしっかり作業できそうですので、早速、データの前処理。まずは必要なデータのみに絞っていきます。
> library(data.table) > dt<-fread("./data/SE.csv") #直近のデータにはタイムとかが入ってないので、削ります。 > dt<-dt[V3<20150721,] > src<-dt[,list(V4,V5,V6,V9,V10,V11,V12,V13,V15,V18,V20,V21,V22,V23,V26,V28,V30,V32,V36,V37,V38,V41,V44,V48,V49,V50,V51,V52,V53,V58,V59,V66,V73)]
ということで、最後に以下のデータ項目に絞り込みを行っています。
| # | ラベル | 項目 |
| 1 | V4 | 開催年 |
| 2 | V5 | 開催月日 |
| 3 | V6 | 競馬場コード |
| 4 | V9 | レース番号 |
| 5 | V10 | 枠番 |
| 6 | V11 | 馬番 |
| 7 | V12 | 血統登録番号 |
| 8 | V13 | 馬名 |
| 9 | V15 | 性別コード |
| 10 | V18 | 馬齢 |
| 11 | V20 | 調教師コード |
| 12 | V21 | 調教師名略称 |
| 13 | V22 | 馬主コード |
| 14 | V23 | 馬主名 |
| 15 | V26 | 負担重量 |
| 16 | V28 | ブリンカー使用区分 |
| 17 | V30 | 騎手コード |
| 18 | V32 | 騎手名略称 |
| 19 | V36 | 馬体重 |
| 20 | V37 | 増減符号 |
| 21 | V38 | 増減差 |
| 22 | V41 | 確定着順 |
| 23 | V44 | 走破タイム |
| 24 | V48 | 1コーナーでの順位 |
| 25 | V49 | 2コーナーでの順位 |
| 26 | V50 | 3コーナーでの順位 |
| 27 | V51 | 4コーナーでの順位 |
| 28 | V52 | 単勝オッズ |
| 29 | V53 | 単勝人気順 |
| 30 | V58 | 後4ハロンタイム |
| 31 | V59 | 後3ハロンタイム |
| 32 | V66 | タイム差 |
| 33 | V73 | 今回レース脚質判定 |
で、これだけだとレース情報がわからないので、レース情報の方も整形していきます。
> dt<-fread("./data/ra.csv") #結果(月曜発表)が出ているレースに絞り込み。 > dt<-dt[V2==7,] > src2<-dt[,list(V4,V5,V6,V9,V10,V11,V12,V18,V23,V25,V28,V29,V30,V31,V32,V33,V34,V36,V40,V41,V42,V43,V44,V60,V62,V65,V66,V67,V94,V95,V96,V97,V98,V100,V101,V103,V104,V106,V107,V109)]
レース情報は以下に絞り込み。多い・・・
| # | ラベル | 項目 | sample |
| 1 | V4 | 開催年 | |
| 2 | V5 | 開催月日 | |
| 3 | V6 | 競馬場コード | |
| 4 | V9 | レース番号 | |
| 5 | V10 | 曜日コード | |
| 6 | V11 | 特別競走番号 | |
| 7 | V12 | 競走名本題 | |
| 8 | V18 | 競走名略称10文字 | |
| 9 | V23 | グレードコード | |
| 10 | V25 | 競争種別コード | |
| 11 | V28 | 競争条件コード2歳条件 | |
| 12 | V29 | 競争条件コード3歳条件 | |
| 13 | V30 | 競争条件コード4歳条件 | |
| 14 | V31 | 競争条件コード5歳条件 | |
| 15 | V32 | 競争条件コード歳若年条件 | |
| 16 | V33 | 競争条件名称 | |
| 17 | V34 | 距離 | |
| 18 | V36 | トラックコード | |
| 19 | V40 | 本賞金1着 | |
| 20 | V41 | 本賞金2着 | |
| 21 | V42 | 本賞金3着 | |
| 22 | V43 | 本賞金4着 | |
| 23 | V44 | 本賞金5着 | |
| 24 | V60 | 発走時刻 | |
| 25 | V62 | 登録頭数 | |
| 26 | V65 | 天候コード | |
| 27 | V66 | 芝馬場状態コード | |
| 28 | V67 | ダート馬場状態コード | |
| 29 | V94 | 前3ハロンタイム | |
| 30 | V95 | 前4ハロンタイム | |
| 31 | V96 | 後3ハロンタイム | |
| 32 | V97 | 後4ハロンタイム | |
| 33 | V98 | コーナー | |
| 34 | V100 | 各通過順位 | |
| 35 | V101 | コーナー | |
| 36 | V103 | 各通過順位 | |
| 37 | V104 | コーナー | |
| 38 | V106 | 各通過順位 | |
| 39 | V107 | コーナー | |
| 40 | V109 | 各通過順位 |
ちょっと欲張りすぎたかな・・・1〜4でレースが一意に決まります。で、今回目的のデータセットを作ります。とりあえず、開催年、開催月日、レース番号、馬番、血統登録番号、馬名、確定着順、単勝人気順、後3ハロンタイム、距離、芝馬場状態コード、ダート馬場状態コードを作ります。
#まずは馬毎レースデータを東京競馬場に絞り込み > data_train <- src[V6==5,] #フィールドの絞り込み > data_train <- data_train[,list(V4,V5,V9,V11,V12,V13,V41,V53,V59)] #続けて開催年・開催月日・レース番号でキーを作成(最初からやっとけよって話ですよね・・・)。 > data_train[,key:=paste(data_train[,V4],data_train[,V5],data_train[,V9])] > data_train <- data_train[,list(key,V11,V12,V13,V41,V53,V59)] #次にレース情報のフィールドを絞り込み #結局(開催年、開催月日、レース番号、レース名称、グレードコード、距離、トラックコード(芝ダ種別)、本賞金1位)にした。 > tmp <- src2[V6==5,] > tmp <- tmp[,list(V4,V5,V9,V18,V23,V34,V36,V40)] #同じくキー作成 > tmp[,key:=paste(tmp[,V4],tmp[,V5],tmp[,V9])] > tmp <- tmp[,list(key,V18,V23,V34,V36,V40)] #で、以下でjoin > install.packages("plyr") > library(plyr) > data_train <- join(data_train,tmp,by="key")
で、さらにここから、各馬のそのレース以前の入賞比率のフィールドを追加します。結構悩みましたが、よくよく考えてみれば、「そのレコード以前のデータの平均」すなわち、株取引でいうところの移動平均じゃん、というところに気がつき、移動平均なら絶対誰かライブラリ作ってるやろ、ってことで探してみました。
・・・と色々探してみましたがコレというものがなく、結局、やりたかった前Nレースの入賞比率ではなく、前全レースの入賞比率を出すことにしました。
#まずは、3着以内フィールドを作成(V41が確定着順)。 > data_train <- data_train[,list(key,V11,V12,V13,V41,ifelse(data_train$V41<4,1,0),V53,V59,V18,V23,V34,V36,V40)] #dplyrをインストール > install.packages("dplyr") > library(dplyr) #説明後述 > data_train<-mutate( group_by(data_train,V12), nyusyo=cummean(V6)) > data_train[V12==2009106253,] Source: local data table [6 x 14] key V11 V12 (略) V13 V41 V6 V53 (略) Variables not shown: V59 (int), V18 (chr), V23 (chr), V34 (int), V36 (int), V40 (int), nyusyo (dbl) #data.frameで出力 > data.frame(data_train[V12==2009106253,]) key V11 V12 (略) V13 V41 V6 V53 V59 (略) V18 V23 V34 V36 V40 nyusyo (略)
ということで、こんな感じです。サラッと書いていますが、これやるだけで数時間ググりまくりました・・・・。
で、結局、かの高名なdplyrを使ってやってみました。よくよく考えたらplyrでもできたきがしますが。data_train<-mutate( group_by(data_train,V12), nyusyo=cummean(V6))では、mutateで各要素について計算した結果を新規列として追加しています。計算式はnyusyo=cummean(V6)です。列ラベルをnyusyoとしたうえで、cummean(V6)の計算結果を入れます。group_byでは、馬コードでグルーピングしています。入賞比率は馬毎に計算しますからね。cummean(V6)ですが、今回一番のミソでしょう。詳細はよくわかりませんが、操作中の要素とそれ以前のすべての要素の平均を出してくれます。ただのmeanだと、全データの平均になってしまいます。ちなみに、cummeanはデフォの関数みたいです・・・すげー探したのに・・・orz
あと、できたデータを出力する時ですが、普通にやるとVariables not shown:が出てしまって後ろの方のフィールドが見えません。コレ調べたんですが、なにやらdplyrの仕様らしいです*1。なので、data.frameにして出力しましょう。
まぁ、とりあえず、求めていたデータセットができました。ですが、はやり東京競馬場だけに限定してしまうとややデータ不足ですね。また、全競馬場含んだものにしようと思います。
ということで、これまでの操作をまとめ。
library(data.table) library(plyr) library(dplyr) #ファイル読み込み dt<-fread("./data/SE.csv") #未更新データ削除 dt<-dt[V3<20150721,] #フィールド絞り込み data_train <- dt[,list(V4,V5,V9,V11,V12,V13,V41,V52,V53,V59)] #開催年・開催月日・レース番号からキー作成 data_train[,key:=paste(data_train[,V4],data_train[,V5],data_train[,V9])] data_train <- data_train[,list(key,V11,V12,V13,V41,V53,V59)] #ファイル読み込み dt<-fread("./data/ra.csv") #未更新データ削除 dt<-dt[V2==7,] #フィールド絞り込み tmp <- dt[,list(V4,V5,V9,V18,V23,V34,V36,V40)] #開催年・開催月日・レース番号からキー作成 tmp[,key:=paste(tmp[,V4],tmp[,V5],tmp[,V9])] tmp <- tmp[,list(key,V18,V23,V34,V36,V40)] #data_trainとtmpをkeyでjoin data_train <- join(data_train,tmp,by="key") #入賞フラグ作成 data_train <- data_train[,list(key,V11,V12,V13,V41,ifelse(data_train$V41<4,1,0),V53,V59,V18,V23,V34,V36,V40)] #入賞比率算出 data_train<-mutate( group_by(data_train,V12), nyusyo=cummean(V6))
軽く分析
ということで、軽くデータに触っていきます。あんまり時間ないのですっげーてきとーです。
> install.packages("data.table") > library(data.table) > system.time(dt<-fread("./data/nkf_SE.csv")) ユーザ システム 経過 1.380 0.324 2.816 #最新のデータははいっていないので除外。 > src<-dt[V3<20150721,] #東京競馬場に絞り込み > tkysrc<-dt[V6==5,] #さらに馬番、確定順位、後3ハロンタイム列に絞り込み、同時に順位が2位以内を1、それ以外を0に。 > umaban_3f<-tkysrc[,list(V11,ifelse(tkysrc$V41<3,1,0),V59)] > head(umaban_3f) V11 V2 V59 1: 1 1 383 2: 2 0 391 3: 3 0 381 4: 4 0 395 5: 5 0 395 6: 6 0 392 > str(umaban_3f) Classes ‘data.table’ and 'data.frame': 43775 obs. of 3 variables: $ V11: int 1 2 3 4 5 6 7 8 9 10 ... $ V2 : num 1 0 0 0 0 0 0 0 0 1 ... $ V59: int 383 391 381 395 395 392 387 389 418 379 ... - attr(*, ".internal.selfref")=<externalptr> > summary(umaban_3f) V11 V2 V59 Min. : 1.00 Min. :0.0000 Min. : 0 1st Qu.: 4.00 1st Qu.:0.0000 1st Qu.:347 Median : 8.00 Median :0.0000 Median :363 Mean : 8.11 Mean :0.1628 Mean :357 3rd Qu.:12.00 3rd Qu.:0.0000 3rd Qu.:380 Max. :18.00 Max. :1.0000 Max. :999 #チェックしたところ、V59に999があるので除外処理。 > umaban_3f<-umaban_3f[V59<999,] > summary(umaban_3f) V11 V2 V59 Min. : 1.000 Min. :0.0000 Min. : 0.0 1st Qu.: 4.000 1st Qu.:0.0000 1st Qu.:347.0 Median : 8.000 Median :0.0000 Median :363.0 Mean : 8.108 Mean :0.1547 Mean :350.8 3rd Qu.:12.000 3rd Qu.:0.0000 3rd Qu.:379.0 Max. :18.000 Max. :1.0000 Max. :954.0
data.tableいいっすね。とりあえず前処理完了。いよいよ分析。
> res<-glm(umaban_3f$V2~.,umaban_3f[,list(V11,V59)],family=binomial) > summary(res) Call: glm(formula = umaban_3f$V2 ~ ., family = binomial, data = umaban_3f[, list(V11, V59)]) Deviance Residuals: Min 1Q Median 3Q Max -1.2346 -0.5516 -0.5109 -0.4668 2.1937 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) 1.2670386 0.0607073 20.871 < 2e-16 *** V11 -0.0091667 0.0031009 -2.956 0.00312 ** V59 -0.0085181 0.0001617 -52.684 < 2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 37357 on 43356 degrees of freedom Residual deviance: 34282 on 43354 degrees of freedom AIC: 34288 Number of Fisher Scoring iterations: 4 > > xp<-seq(min(umaban_3f[,V11]),max(umaban_3f[,V11]),0.1) > yp<-seq(min(umaban_3f[,V59]),max(umaban_3f[,V59]),0.1) > grid<-expand.grid(V11=xp,V59=yp) > head(grid) V11 V59 1 1.0 0 2 1.1 0 3 1.2 0 4 1.3 0 5 1.4 0 6 1.5 0 > length(xp) [1] 171 > Z<-predict(res,grid,type="response") > plot(umaban_3f[,V11],umaban_3f[,V59],col=ifelse(umaban_3f[,V2],"red","blue")) > contour(xp,yp,matrix(Z,length(xp)),add=T,levels=0.5,col="green")

ありゃ、、まぁ距離で分けてないからタイムで別れるのは当然か。。。しかし、0は除外しないとな。。
ということで、短距離について再度トライ。
> umaban_3f_short<-umaban_3f[V59<200,] > umaban_3f_short<-umaban_3f_short[V59>0,] > summary(umaban_3f_short) V11 V2 V59 Min. : 1.00 Min. :0.0000 Min. :130.0 1st Qu.: 4.00 1st Qu.:0.0000 1st Qu.:135.0 Median : 7.00 Median :0.0000 Median :137.0 Mean : 7.33 Mean :0.1562 Mean :137.5 3rd Qu.:11.00 3rd Qu.:0.0000 3rd Qu.:139.0 Max. :14.00 Max. :1.0000 Max. :155.0 > > res2<-glm(umaban_3f_short$V2~.,umaban_3f_short[,list(V11,V59)],family=binomial) > summary(res2) Call: glm(formula = umaban_3f_short$V2 ~ ., family = binomial, data = umaban_3f_short[, list(V11, V59)]) Deviance Residuals: Min 1Q Median 3Q Max -1.2915 -0.6271 -0.4517 -0.2113 2.8943 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) 46.57868 4.78751 9.729 <2e-16 *** V11 -0.03023 0.01937 -1.560 0.119 V59 -0.35216 0.03527 -9.985 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 1253.9 on 1446 degrees of freedom Residual deviance: 1110.4 on 1444 degrees of freedom AIC: 1116.4 Number of Fisher Scoring iterations: 5 > > xp<-seq(min(umaban_3f_short[,V11]),max(umaban_3f_short[,V11]),0.1) > yp<-seq(min(umaban_3f_short[,V59]),max(umaban_3f_short[,V59]),0.1) > grid<-expand.grid(V11=xp,V59=yp) > length(xp) [1] 131 > Z<-predict(res2,grid,type="response") > plot(umaban_3f_short[,V11],umaban_3f_short[,V59],col=ifelse(umaban_3f_short[,V2],"red","blue")) > contour(xp,yp,matrix(Z,length(xp)),add=T,levels=0.5,col="green")
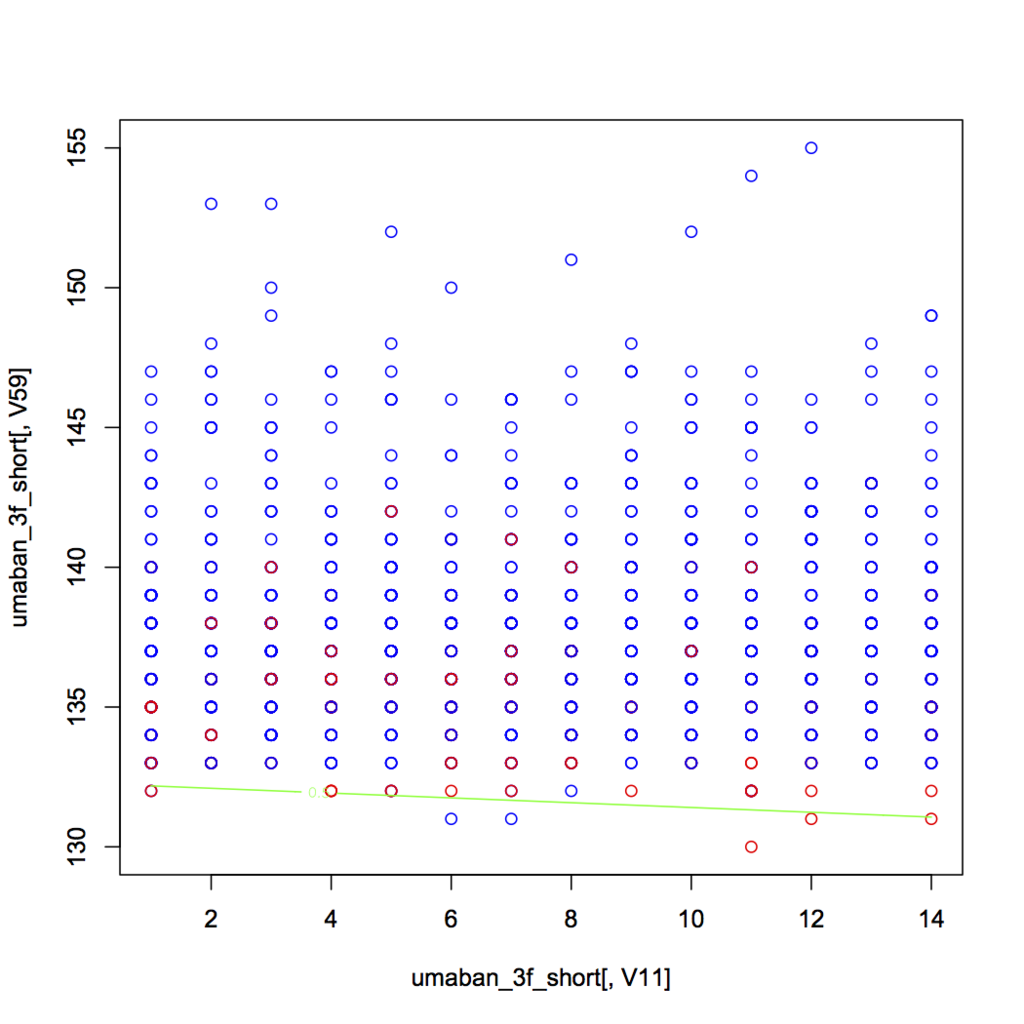
うーん、なんか全然ダメだな。てか、そもそも馬番入れたのがまずかったか・・・でも、この中枠の奴らが3f早いけど入賞してないってのはミクロに見ていくと面白いかも。ということで、次は単純にlmやる。
> rank_3f<-tkysrc[,list(V41,V59)] > head(rank_3f) V41 V59 1: 1 383 2: 8 391 3: 9 381 4: 10 395 5: 13 395 6: 6 392 > rank_3f<-rank_3f[V59<200,] > rank_3f<-rank_3f[V59>0,] > head(rank_3f[,V59]) [1] 137 136 143 139 137 140 > length(rank_3f[,V59]) [1] 1447 > res3<-lm(rank_3f[,V41]~rank_3f[,V59]) > summary(res3) Call: lm(formula = rank_3f[, V41] ~ rank_3f[, V59]) Residuals: Min 1Q Median 3Q Max -8.5462 -2.5187 0.0017 2.3922 8.4813 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) -72.1541 3.4389 -20.98 <2e-16 *** rank_3f[, V59] 0.5754 0.0250 23.01 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 3.239 on 1445 degrees of freedom Multiple R-squared: 0.2682, Adjusted R-squared: 0.2677 F-statistic: 529.6 on 1 and 1445 DF, p-value: < 2.2e-16 > plot(rank_3f[,V59],rank_3f[,V41]) > abline(a=-72.1541,b=0.5754,col="red",lty=2)
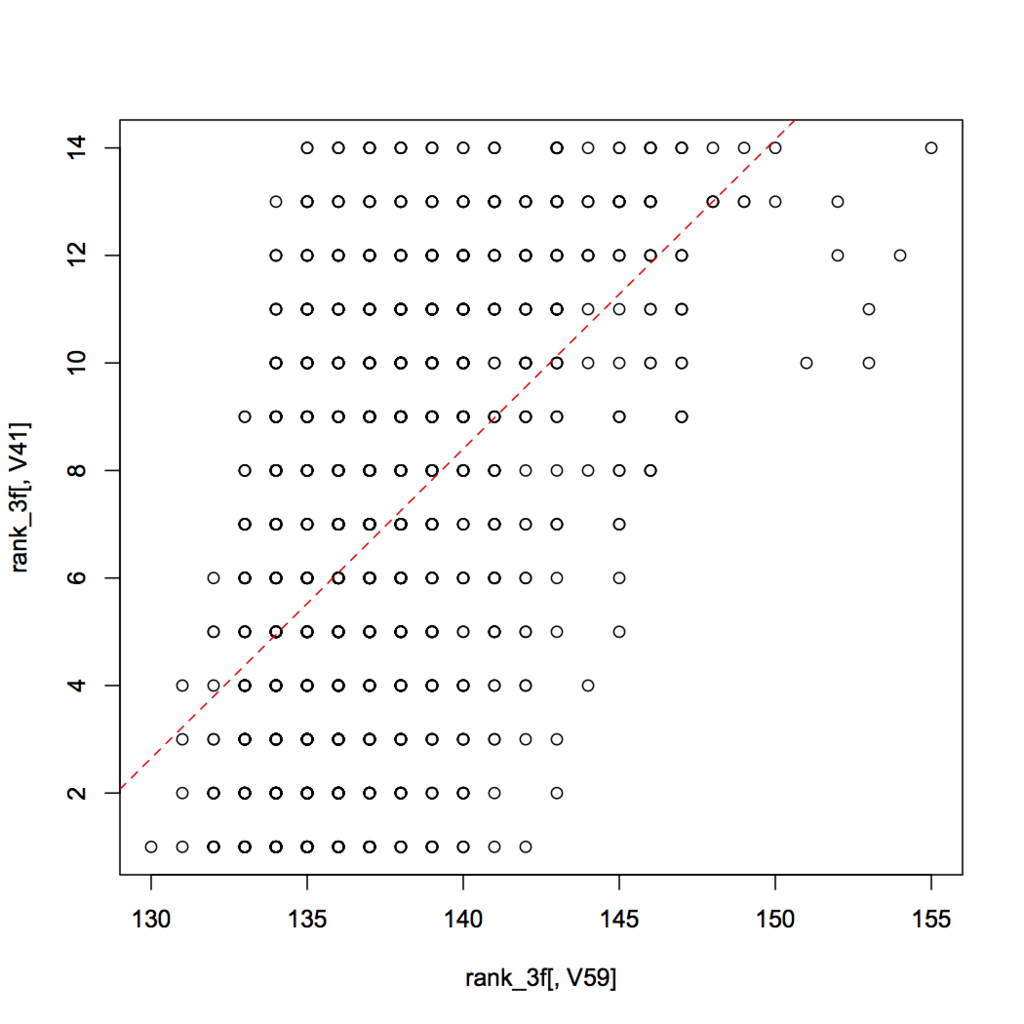
あーんー、そーだよね。こーなるよね。まぁこれも後3ハロンタイムが速いほど、そのまま上位になりやすいってのはわかるけど、やっぱ来週はちゃんと腰を据えてやろう。。
やりたいこと
公営競技の統計学的アプローチによる分析および最適投票合議システムの実装
つまるところ、お馬さんの分析ですよ。そうです、これをやりたいがためにココまで練習してきたんです。
ということで、ここまで準備してきたものを使って簡単なシステムを作ってみたいと思います。やっぱり作るならサクッとお金になるのがじっくり研究しがいのあるテーマがいいよねー、ということで、元々お馬さん大好きなこともあり、研究テーマとして自ずと本テーマを選択していました。漢の夢ですよね。
実は、ここ半年、無駄に長い通勤時間をフルに使ってずっとアプローチを考えていました。
実装する(予定)の統計学的アプローチ
はい、「統計学的アプローチ」って言いたいだけです。頭良さそうですよね笑
で、最初は簡単なところから攻めていきたいと思います。
- ロジスティック回帰分析
- SVM
- Random Forest
- NN
- オリジナル指数分析(スコアリング)
- その他の分析
- 血統分析 etc.
で、統計素人でもある私はここ数ヶ月必死にググりまくってましたが、以下のサイトがとてもわかりやすくザクッと説明が書いてあるので、入り口として非常に助かりました(ありがとうございました)。なんの分野でも、こーゆー感じで素人にもわかりやすく説明できるようになりたいですよね。
回帰分析とその応用① ~回帰分析は何のために行うのか? | GiXo Ltd.
分析の練習(2)
ロジスティック回帰分析
irisのデータを使って練習。
irisは
> head(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 4 4.6 3.1 1.5 0.2 setosa 5 5.0 3.6 1.4 0.2 setosa 6 5.4 3.9 1.7 0.4 setosa
って感じで、1:50がsetosa、51:100がversicolor、101:150がvirginicaとなっている。今回はロジスティック回帰ということで、51:150に絞ってやる。また、変数もSepal.LengthとPetal.Lengthの二つに絞る。
> test <- iris[51:150,c(1,3,5)] > test$Species<-ifelse(test$Species=="versicolor",1,0) > head(test) Sepal.Length Petal.Length Species 51 7.0 4.7 1 52 6.4 4.5 1 53 6.9 4.9 1 54 5.5 4.0 1 55 6.5 4.6 1 56 5.7 4.5 1 > > > res<-glm(test$Species~.,test[1:2],family=binomial) > summary(res) Call: glm(formula = test$Species ~ ., family = binomial, data = test[1:2]) Deviance Residuals: Min 1Q Median 3Q Max -1.54924 -0.02909 0.00001 0.04669 2.81936 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) 39.839 13.089 3.044 0.002338 ** Sepal.Length 4.017 1.623 2.474 0.013348 * Petal.Length -13.313 3.913 -3.402 0.000669 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 138.63 on 99 degrees of freedom Residual deviance: 23.85 on 97 degrees of freedom AIC: 29.85 Number of Fisher Scoring iterations: 9
こんな感じ。次はできた直線をplotしていく。
> xp<-seq(min(test[,1]),max(test[,1]),0.1) > yp<-seq(min(test[,2]),max(test[,2]),0.1) > cushT<-expand.grid(Sepal.Length=xp,Petal.Length=yp) #x軸(Sepal.Length)、y軸(Petal.Length)について0.1刻みのgridを作る。expand.gridではラベル名を指定。 > head(cushT) Sepal.Length Petal.Length 1 4.9 3 2 5.0 3 3 5.1 3 4 5.2 3 5 5.3 3 6 5.4 3 > length(xp) [1] 31 #0.1刻みで要素数が31個 > Z<-predict(res,cushT,type="response") #作ったgridの全要素に対して評価。Zには結果が一次配列で格納される。 > plot(test[,1],test[,2],col=ifelse(test[,3],"red","blue")) #もともとのデータをplot > contour(xp,yp,matrix(Z,length(xp)),add=T,levels=0.5,col="green") #評価結果をplot。
最後のcontourが分かりにくいが、今回は、z=ax+byという関数を二次元でplotしようとしているため、zをどうにか表現しないといけない。そこで、contourを用いる。この関数は等高線を引くための関数で、matrix(Z,length(xp))でlength(xp)×length(xy)の全要素の高さZを指定してあげて、levels=0.5の線を引く、という感じ。

SVM
続いて、SVM(Support Vector Machine)。
パッケージとしては、kernlabってのもあるみたいだけど、なんかエラーが出てインストールできなかったので、e1071でトライ。
とりあえずインストールした後、irisの半分を訓練データとして確保し、残りを評価する。
> install.packages("e1071") > library(e1071) > rowdata<-nrow(iris) > random_ids<-sample(rowdata,rowdata*0.5) > random_ids [1] 2 111 107 143 122 119 121 114 10 84 103 6 60 109 11 118 138 89 113 [20] 88 75 129 76 125 91 15 64 140 80 36 74 52 145 47 102 135 94 13 [39] 82 142 23 8 33 71 55 69 85 133 58 37 81 97 56 16 137 7 131 [58] 42 28 100 53 104 9 87 26 27 95 120 90 66 30 83 25 77 61 > iris_training<-iris[random_ids,] > iris_training Sepal.Length Sepal.Width Petal.Length Petal.Width Species 2 4.9 3.0 1.4 0.2 setosa 111 6.5 3.2 5.1 2.0 virginica 107 4.9 2.5 4.5 1.7 virginica 143 5.8 2.7 5.1 1.9 virginica 122 5.6 2.8 4.9 2.0 virginica > iris_predicting<-iris[-random_ids,] > iris_predicting Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 4 4.6 3.1 1.5 0.2 setosa 5 5.0 3.6 1.4 0.2 setosa 12 4.8 3.4 1.6 0.2 setosa 14 4.3 3.0 1.1 0.1 setosa 17 5.4 3.9 1.3 0.4 setosa 18 5.1 3.5 1.4 0.3 setosa 19 5.7 3.8 1.7 0.3 setosa > iris_svm<-svm(Species~.,data=iris_training) > iris_svm Call: svm(formula = Species ~ ., data = iris_training) Parameters: SVM-Type: C-classification SVM-Kernel: radial cost: 1 gamma: 0.25 Number of Support Vectors: 38 > result_predict<-predict(iris_svm,iris_predicting) > result_predict 1 3 4 5 12 14 17 setosa setosa setosa setosa setosa setosa setosa 18 19 20 21 22 24 29 setosa setosa setosa setosa setosa setosa setosa 31 32 34 35 38 39 40 setosa setosa setosa setosa setosa setosa setosa 41 43 44 45 46 48 49 setosa setosa setosa setosa setosa setosa setosa 50 51 54 57 59 62 63 setosa versicolor versicolor versicolor versicolor versicolor versicolor 65 67 68 70 72 73 78 versicolor versicolor versicolor versicolor versicolor versicolor versicolor 79 86 92 93 96 98 99 versicolor versicolor versicolor versicolor versicolor versicolor versicolor 101 105 106 108 110 112 115 virginica virginica virginica virginica virginica virginica virginica 116 117 123 124 126 127 128 virginica virginica virginica virginica virginica virginica virginica 130 132 134 136 139 141 144 virginica virginica versicolor virginica virginica virginica virginica 146 147 148 149 150 virginica virginica virginica virginica virginica Levels: setosa versicolor virginica > table(result_predict,iris_predicting$Species) result_predict setosa versicolor virginica setosa 29 0 0 versicolor 0 20 1 virginica 0 0 25 > (29+20+25)/75 [1] 0.9866667
ということで、irisデータでは98.66%の精度に!なるほどよくわからんがすごそうだな
分析の練習
R環境が整ったところで、そろそろ実際の分析の練習をしてみようと思います。
Rにはすでにいろんなデータが入っているので、それらを使ってやってみようと思います。
まずは、どんなデータが入っているのか確認。
> data() Data sets in package ‘datasets’: AirPassengers Monthly Airline Passenger Numbers 1949-1960 BJsales Sales Data with Leading Indicator BJsales.lead (BJsales) Sales Data with Leading Indicator BOD Biochemical Oxygen Demand CO2 Carbon Dioxide Uptake in Grass Plants ChickWeight Weight versus age of chicks on different diets DNase Elisa assay of DNase EuStockMarkets Daily Closing Prices of Major European Stock Indices, 1991-1998 Formaldehyde Determination of Formaldehyde HairEyeColor Hair and Eye Color of Statistics Students Harman23.cor Harman Example 2.3 Harman74.cor Harman Example 7.4 Indometh Pharmacokinetics of Indomethacin InsectSprays Effectiveness of Insect Sprays JohnsonJohnson Quarterly Earnings per Johnson & Johnson Share LakeHuron Level of Lake Huron 1875-1972 LifeCycleSavings Intercountry Life-Cycle Savings Data Loblolly Growth of Loblolly pine trees Nile Flow of the River Nile Orange Growth of Orange Trees OrchardSprays Potency of Orchard Sprays PlantGrowth Results from an Experiment on Plant Growth Puromycin Reaction Velocity of an Enzymatic Reaction Seatbelts Road Casualties in Great Britain 1969-84 Theoph Pharmacokinetics of Theophylline Titanic Survival of passengers on the Titanic ToothGrowth The Effect of Vitamin C on Tooth Growth in Guinea Pigs UCBAdmissions Student Admissions at UC Berkeley UKDriverDeaths Road Casualties in Great Britain 1969-84 UKgas UK Quarterly Gas Consumption USAccDeaths Accidental Deaths in the US 1973-1978 USArrests Violent Crime Rates by US State USJudgeRatings Lawyers' Ratings of State Judges in the US Superior Court USPersonalExpenditure Personal Expenditure Data UScitiesD Distances Between European Cities and Between US Cities VADeaths Death Rates in Virginia (1940) WWWusage Internet Usage per Minute WorldPhones The World's Telephones ability.cov Ability and Intelligence Tests airmiles Passenger Miles on Commercial US Airlines, 1937-1960 airquality New York Air Quality Measurements anscombe Anscombe's Quartet of 'Identical' Simple Linear Regressions attenu The Joyner-Boore Attenuation Data attitude The Chatterjee-Price Attitude Data austres Quarterly Time Series of the Number of Australian Residents beaver1 (beavers) Body Temperature Series of Two Beavers beaver2 (beavers) Body Temperature Series of Two Beavers cars Speed and Stopping Distances of Cars chickwts Chicken Weights by Feed Type co2 Mauna Loa Atmospheric CO2 Concentration crimtab Student's 3000 Criminals Data discoveries Yearly Numbers of Important Discoveries esoph Smoking, Alcohol and (O)esophageal Cancer euro Conversion Rates of Euro Currencies euro.cross (euro) Conversion Rates of Euro Currencies eurodist Distances Between European Cities and Between US Cities faithful Old Faithful Geyser Data fdeaths (UKLungDeaths) Monthly Deaths from Lung Diseases in the UK freeny Freeny's Revenue Data freeny.x (freeny) Freeny's Revenue Data freeny.y (freeny) Freeny's Revenue Data infert Infertility after Spontaneous and Induced Abortion iris Edgar Anderson's Iris Data iris3 Edgar Anderson's Iris Data islands Areas of the World's Major Landmasses ldeaths (UKLungDeaths) Monthly Deaths from Lung Diseases in the UK lh Luteinizing Hormone in Blood Samples longley Longley's Economic Regression Data lynx Annual Canadian Lynx trappings 1821-1934 mdeaths (UKLungDeaths) Monthly Deaths from Lung Diseases in the UK morley Michelson Speed of Light Data mtcars Motor Trend Car Road Tests nhtemp Average Yearly Temperatures in New Haven nottem Average Monthly Temperatures at Nottingham, 1920-1939 npk Classical N, P, K Factorial Experiment occupationalStatus Occupational Status of Fathers and their Sons precip Annual Precipitation in US Cities presidents Quarterly Approval Ratings of US Presidents pressure Vapor Pressure of Mercury as a Function of Temperature quakes Locations of Earthquakes off Fiji randu Random Numbers from Congruential Generator RANDU rivers Lengths of Major North American Rivers rock Measurements on Petroleum Rock Samples sleep Student's Sleep Data stack.loss (stackloss) Brownlee's Stack Loss Plant Data stack.x (stackloss) Brownlee's Stack Loss Plant Data stackloss Brownlee's Stack Loss Plant Data state.abb (state) US State Facts and Figures state.area (state) US State Facts and Figures state.center (state) US State Facts and Figures state.division (state) US State Facts and Figures state.name (state) US State Facts and Figures state.region (state) US State Facts and Figures state.x77 (state) US State Facts and Figures sunspot.month Monthly Sunspot Data, from 1749 to "Present" sunspot.year Yearly Sunspot Data, 1700-1988 sunspots Monthly Sunspot Numbers, 1749-1983 swiss Swiss Fertility and Socioeconomic Indicators (1888) Data treering Yearly Treering Data, -6000-1979 trees Girth, Height and Volume for Black Cherry Trees uspop Populations Recorded by the US Census volcano Topographic Information on Auckland's Maunga Whau Volcano warpbreaks The Number of Breaks in Yarn during Weaving women Average Heights and Weights for American Women
いやまぁたくさんはいってますね。
ということで、今回はよく使われているairqualityを使いたいと思います。
> head(airquality) Ozone Solar.R Wind Temp Month Day 1 41 190 7.4 67 5 1 2 36 118 8.0 72 5 2 3 12 149 12.6 74 5 3 4 18 313 11.5 62 5 4 5 NA NA 14.3 56 5 5 6 28 NA 14.9 66 5 6 > str(airquality) 'data.frame': 153 obs. of 6 variables: $ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ... $ Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ... $ Wind : num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ... $ Temp : int 67 72 74 62 56 66 65 59 61 69 ... $ Month : int 5 5 5 5 5 5 5 5 5 5 ... $ Day : int 1 2 3 4 5 6 7 8 9 10 ...
head()はそのデータの最初の方をチラ見する関数ですね。str()ってのはstructure、データの構造を見るやつらしいです。ふむふむ。
で、このデータを使っていろいろやってきます。
まず、Rすげーってなるよしなに関数からいきます。
- plot()
> plot(airquality)

おおお!これがplot()の真価・・・すげーそれっぽい・・・これだけ見てもおもしろいですね〜
- summary()
> summary(airquality) Ozone Solar.R Wind Temp Min. : 1.00 Min. : 7.0 Min. : 1.700 Min. :56.00 1st Qu.: 18.00 1st Qu.:115.8 1st Qu.: 7.400 1st Qu.:72.00 Median : 31.50 Median :205.0 Median : 9.700 Median :79.00 Mean : 42.13 Mean :185.9 Mean : 9.958 Mean :77.88 3rd Qu.: 63.25 3rd Qu.:258.8 3rd Qu.:11.500 3rd Qu.:85.00 Max. :168.00 Max. :334.0 Max. :20.700 Max. :97.00 NA's :37 NA's :7 Month Day Min. :5.000 Min. : 1.0 1st Qu.:6.000 1st Qu.: 8.0 Median :7.000 Median :16.0 Mean :6.993 Mean :15.8 3rd Qu.:8.000 3rd Qu.:23.0 Max. :9.000 Max. :31.0
おおーー、もうこれでいいじゃんって感じですね。
でだ、早速、分析っぽいことをやってみようと思う。まずは単回帰分析。
はて、、という感じですが、今回は、オゾン量?と気温の相関を調べます。
なんか、回帰分析ってのは、「1つの目的変数を1つの説明変数で予測する手法」だそうで、ぶっちゃけ簡単に言うと、
目的変数(オゾン量)=定数1×説明変数(気温)+変数2
って形で表す感じで、過去のデータから線形グラフを求めて、未来のデータを予測するというものだそうです。
で、説明変数が複数になると「重回帰分析」と呼ぶそう。
-P値 ちなみに、定数のことを「偏回帰係数」と言います。ただ、偏回帰係数はそれぞれの変数が目的変数に どの程度影響を与えるか、ってのを直接表すものじゃない(cmをmにすると変わるし)ってことで、 じゃ、影響度合いを調べる指標ないのよ?ってことで、「P値」があります。詳細は省略しますが、 だいたい5%未満であれば、その係数は統計的に意味があると言えるそうです。 -マルチコ(多重共線性) 重回帰分析の場合、説明変数同士が高い相関関係にある場合、分析結果が不安定になるそうです。 これをマルチコリニアリティ(multicollinearity)と言います。なので、重回帰分析をする場合は、 各説明変数同士の相関関係を調べてからやったほうがいいみたいですね。具体的には、説明変数同士の 相関係数が0.5以上なら注意、0.7以上ならNGだそうです。
ということで、前振りが長くなりましたが、いざ分析。
オゾン量を気温で説明します。ハイ。
> cor(airquality$Ozone,airquality$Temp,use="complete.obs") [1] 0.6983603 > result <- lm(Ozone~Temp,data=airquality) > summary(result) Call: lm(formula = Ozone ~ Temp, data = airquality) Residuals: Min 1Q Median 3Q Max -40.729 -17.409 -0.587 11.306 118.271 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) -146.9955 18.2872 -8.038 9.37e-13 *** Temp 2.4287 0.2331 10.418 < 2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 23.71 on 114 degrees of freedom (37 observations deleted due to missingness) Multiple R-squared: 0.4877, Adjusted R-squared: 0.4832 F-statistic: 108.5 on 1 and 114 DF, p-value: < 2.2e-16
まずは、cor()でオゾン量と気温の相関を求めています(use="complete.obs"はNAの時の扱い。この場合は、NAのある項目は排除する設定。)
で、相関が0.7くらいなのでそこそこの正の相関があるようです。つまり、分析する価値あり。
で、lmってのが回帰分析の関数らしいです。
これで、分析した結果をsummaryします。
結果の真ん中あたりのCoefficients:に偏回帰係数(Estimate)が出てます。つまり、
目的変数(オゾン量)=2.429×説明変数(気温)-146.995
ってことですね。
(ちなみに、Coefficientsの後ろの方の*が多いほど、有意らしいです。MAX星3つなので、気温変数は三ツ星獲得です!
で、これで線形のグラフがかけますので、plotしてみます。
> plot(b,a) > abline(a=-146,b=2.4,col="red",lty=2)

いや〜素敵ですね笑
なんかそれっぽいじゃないっすか!
はじめてのR(2)
ということで、無事Rを入れられたので、次はRubyでRを使ってみる。
調べてみると、いくつか方法があるみたいで、
- RSRuby
- RinRuby
- Rserve
が主流のよう。いろいろググってみた感じで、今回はRSRubyにすることにしました。
インストール
ちなみに環境は以下。
$ sw_vers ProductName: Mac OS X ProductVersion: 10.10.4 BuildVersion: 14E46 $ ruby -v ruby 2.0.0p481 (2014-05-08 revision 45883) [universal.x86_64-darwin14] $ r -q > version _ platform x86_64-apple-darwin14.3.0 arch x86_64 os darwin14.3.0 system x86_64, darwin14.3.0 status major 3 minor 2.1 year 2015 month 06 day 18 svn rev 68531 language R version.string R version 3.2.1 (2015-06-18) nickname World-Famous Astronaut
ということで、インストール。
といきたいところだったけど、どうやらincludeパスとlibパスをちゃんと設定してあげないといけないらしく、しかも、だいたい/Library/Frameworks/R.framework/だって書いてあるけど、そこにはなにもなく、、、
(/Library/Frameworks以下って自分でつくんのかな?素人すぎてわかりません・・・てか、この辺の使い方てきとーすぎて間違ってる気がしてならない・・・笑)
まぁとりあえずパスをちゃんと指定すればいいんだろ〜ってことで、R先生がどこにいるか調べる。
$ brew info R homebrew/science/R: stable 3.2.1 (bottled), HEAD http://www.r-project.org/ /usr/local/Cellar/R/3.2.1_1 (2172 files, 59M)
/usr/local/Cellar/Rにいるってよ。って、brewがそういう設定だから当たり前か。
で、Rディレクトリ以下を見てみると、R.frameworkがいたので、これ指定してあげれば動くでしょ、ってことで以下インストール。
$ sudo gem install rsruby -- --with-R-include=/usr/local/Cellar/r/3.2.1_1/R.framework/Headers --with-R-lib=/usr/local/Cellar/r/3.2.1_1/R.framework/Libraries
で、なにやらR_HOMEをちゃんと指定してあげてねってことなので、.bash_profileもお手入れ。
$ vi ~/.bash_profile # ↓を追記 export R_HOME=/usr/local/Cellar/r/3.2.1_1/R.framework/Resources $ source ~/.bash_profile
ようやくインストール完了。